Die schlechte Nachricht: es ist eine Programmiersprache! Dies wird einen Großteil der Studenten etc. erstmal abschrecken, aber sich mit R zu beschäftigen lohnt sich!
R ist eine Programmiersprache speziell für Datenanalyse und Grafiken. Aus diesem Grund stehen mächtige Funktionen bereits fertig implementiert zur Verfügung. Und das ist der riesige Vorteil einer Programmiersprache: man muss sich nicht in Menüs rumschlagen und ist nicht darauf beschränkt, was in einem Programm als Funktion vorgesehen ist. Besonders bei sich wiederholenden Abläufen ist eine Programmiersprache einfach um einiges schneller. Man muss dabei auch nicht die komplette Sprache lernen, weil es ausreicht, sich die Befehle einzuprägen, die man häufig benötigt und lernt, wo man Hilfe bekommt, falls man vor neuen Herausforderungen steht.
Die Möglichkeiten mit R zu arbeiten
Interaktive R-Konsole
R ist eine Programmiersprache und deshalb ist die bevorzugte Arbeitsweise eine Konsole, wo interaktiv Befehle an R gesendet werden können. Dieser interaktive Modus eignet sich hervorragend, wenn man R nur schnell als statistischen Taschenrechner verwenden möchte. Um eine interaktive R-Konsole zu starten, rufen Sie im Terminal einfach mit
[nutzer@computer ~] \) R
die Programmierumgebung von R auf. Es folgen Informationen zur Version etc. und schon wartet R auf Eingabe.
Am Eingabeprompt von R (>) können nun interaktiv Befehle an R gesendet werden. Nutzen Sie R zum Beispiel als Taschenrechner:
> 2 + 2 [1] 4 > cos(2*pi) [1] 1
Graphisches Frontend für R
Für R existieren auch verschiedene grafische Benutzeroberflächen, jedoch ist R vorwiegend eine Programmiersprache und spielt erst hier ihre wahren Stärken aus. Aber gerade als Anfänger fühlt man sich in einer GUI sicherer, vor allem, wenn man die Befehle noch nicht kennt. Eine GUI ist zum Beispiel Rcmdr. Um die Oberfläche Rcmdr zu verwenden, müssen Sie im Terminal eine interaktive R-Konsole starten. Geben Sie dann am Eingabeprompt von R folgenden Befehl ein:
> library("Rcmdr")
und R startet den R-Commander.
RStudio
Rstudio ist eine neue Entwicklung und sieht sehr vielversprechend aus (Webseite). Mit fertigen Packeten fuer Windows, Mac und Linux die wahrscheinlich vollstaendigste grafische Arbeitsumgebung zur Zeit erhaeltlich.
Grundlagen der Programmierung mit R
R als Taschenrechner
Wie bereits kurz angedeutet wurde, kann man R auch als mächtigen Taschenrechner verwenden. Folgende Übersicht gibt einen Einblick in die Möglichkeiten:

Zuweisungen
Um Werte und Ergebnisse weiterverwenden zu können, muss man sie einem Objekt zuordnen können. Dies geschieht ganz einfach mit
> x1 <- cos(pi/2) # dem Objekt x1 wird ein Wert zugeordnet
> x1 # Ausgeben des Wertes
[1] 6.123234e-17 # Eine typische Merkwürdigkeit der Numerik am
Computer
> round(x1) # weiterverwenden eines Objektes
[1] 0
Werte lassen sich also mit <- einem Objekt zuordnen. Wer bereits mit anderen Programmiersprachen gearbeitet hat, wird diese Art der Zuweisung vielleicht etwas komisch finden, aber wenn man seinen Code sauber schreibt, ist er durch den "Pfeil" logisch und gut lesbar.
Objektnamen sollten am besten mit einem Buchstaben beginnen und R beachtet Groß- und Kleinschreibung! Wenn ein Objektname Sonderzeichen etc. enthält, ist der Zugriff schwierig, jedoch mit Backticks möglich (Bsp: `1x`)
> x1 <- 2 > X1 <- 3 > x1 == X1 # R unterscheidet Groß- und Kleinschreibung [1] FALSE
Ein Objekt besitzt immer einen Datentyp, welcher sich mit mode() abfragen lässt.
> x1 <- 1 > x2 <- "Eins" > mode(x1) [1] "numeric" > mode(x2) [1] "character"
Objekte können aber noch zusätzliche Attribute besitzen, was zum Beispiel bei einer Matrix einleuchtet. So besitzt die Matrix im folgenden Beispiel die Dimension 4x3:
> x1 <- matrix(1:12,4)
> x1
[,1] [,2] [,3]
[1,] 1 5 9
[2,] 2 6 10
[3,] 3 7 11
[4,] 4 8 12
> mode(x1)
[1] "numeric"
> attributes(x1)
$dim
[1] 4 3
Wie man Hilfe bekommt
R besitzt ein integriertes Hilfesystem, was sich zum Beispiel mit der Funktion help() aufrufen lässt. So können Sie sich ja zum Beispiel mal die Hilfeseite zu den Winkelfunktionen anschauen:
> help(cos)
in der Standardeinstellung wird die Hilfe als formatierter Text in R angezeigt. Um die Hilfe zu beenden drücken Sie q Eine weitere Möglichkeit die Hilfe aufzurufen ist mit ? vor dem Funktionsnamen, für den man Hilfe benötigt:
> ?cos
Es lässt sich die Hilfe auch durchsuchen, was nützlich ist, wenn man den genauen Funktionsnamen nicht kennt.
> help.search("Suchbegriff")
> apropos("cos") # mit einem Teil nach dem Namen einer
Funktion suchen
[1] "acos" "acosh" "cos" "cosh"
Mit Hilfe des Befehls help.start() lässt sich eine komfortable HTML Hilfe im Browser starten.
Eine Beispielsitzung
Mit der R Grundinstallation wird auch der berühmte Datensatz von Fisher und Anderson zu den Sepalen und Petalen von 3 Iris Species zur Verfügung gestellt.
> ?iris # gibt Auskunft zum Iris-Datensatz
> iris # zeigt den kompletten Datensatz an
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
6 5.4 3.9 1.7 0.4 setosa
7 4.6 3.4 1.4 0.3 setosa
...
> summary(iris) # zeigt eine Zusammenfassung mit Hilfe
beschreibender Statistik
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
Min. :4.300 Min. :2.000 Min. :1.000 Min. :0.100 setosa :50
1st Qu.:5.100 1st Qu.:2.800 1st Qu.:1.600 1st Qu.:0.300 versicolor:50
Median :5.800 Median :3.000 Median :4.350 Median :1.300 virginica :50
Mean :5.843 Mean :3.057 Mean :3.758 Mean :1.199
3rd Qu.:6.400 3rd Qu.:3.300 3rd Qu.:5.100 3rd Qu.:1.800
Max. :7.900 Max. :4.400 Max. :6.900 Max. :2.500
> attach(iris) # die Namen der Variablen der Irisdaten laden
> Species # Variable Species anzeigen
> spec.num <- as.numeric(Species) # Namen in Zahlen umwandeln und in
> spec.num Objekt spec.num speichern und anzeigen
> plot(Sepal.Length, col=spec.num) # Scatterplot der Sepal.Length
mit verschiedenen Farben col=
> plot(iris[ ,1], iris[ ,3], col=spec.num) # Plot Sepal.Length gegen Petal.Length
> hist(Sepal.Length) # ein einfaches Histogramm
> boxplot(Sepal.Length ~ Species, main=colnames(iris)[1]) # ein Boxplot
Erklärung der verwendeten Befehle
- ?: Aufrufen der Hilfe, beenden mit der Taste q
- summary() Zusammenfassung eines Datensatzes mit Hilfe beschreibender Statistik
-
plot() Sehr umfangreiche Funktion zum Erstellen verschiedener Plots. Dabei ist das Ergebnis abhängig vom Typ der darzustellenden Daten. Der allgemeine Aufruf ist plot(x,y) Wird nur eine variable an plot() übergeben, wird ein Scatterplot gegen den Index des Datensatzes erstellt. (siehe Beispielsitzung).
- Es existieren verschiedene optionale Argumente:
- col= Einstellungen zur Farbe der Datenpunkte
- type= Typ des Plots (Punkte, Linien, etc.)
- main= Titel
- sub= Untertitel
- xlab= x-Achsenbeschriftung
- ylab = y-Achsenbeschriftung
- Für eine ausführliche Hilfe einfach ?plot eingeben und ein wenig experimentieren.
- hist() Erstellen eines Histogramms. Für die optionalen Argumente siehe Hilfe: ?hist
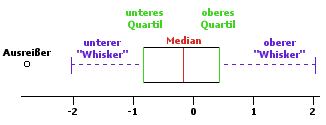
- boxplot() Der Boxplot stellt verschiedene Streuungsmaße der Stichprobe dar. Die Größe der Box umspannt dabei 50% der Daten. Die Box wird vom unteren und vom oberen Quartil begrenzt. Ein Quartil umfasst dabei \( \frac{1}{4} \) der Daten. Das untere Quartil umfasst dabei \( Q_{.25} = \frac{1}{4}\), das obere Quartil \( Q_{.75} = \frac{3}{4} \) der Daten. Der Median entspricht dem mittleren Quartil und damit \( Q_{.5}=\frac{1}{2} \) der Daten. Die Position des Medians innerhalb der Box gibt dabei einen Eindruck über die Schiefe der Daten. Die Whisker umfassen bei R 95% der Daten. Extremere Werte werden als Ausreißer eingetragen. Mit Hilfe des Parameters notch=TRUE wird die Box um den Mittelwert nach der Formel \( \pm 1.58 \frac{IQR}{\sqrt{n}} \) eingeschnürt. \( IQR \) ist der Interquantileabstad und damit die Länge der Box. Das gibt näherungsweise das 95% Koinfidenzinterval des Mittelwertes an. Wenn bei zwei Boxplots sich diese Einschnürungen nicht überschneiden, ist der Unterschied beider Mediane signifikant.
Ein weiteres Beispiel
> set.seed(1) # Reproduzieren von Zufallszahlen
> x <- rnorm(1000) # Ziehen von 1000 standardnormalverteilten
# Zufallszahlen
# Histogramm mit theoretischer Dichtefunktion und Legende
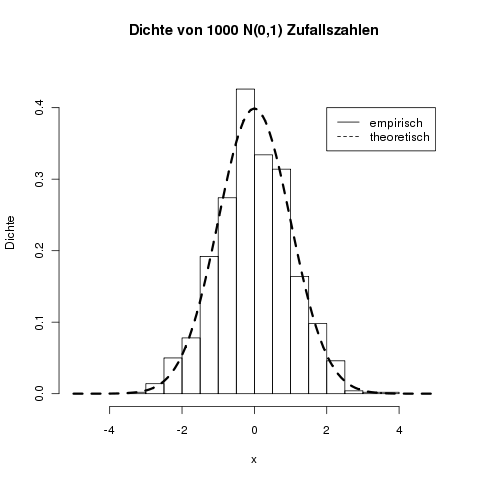
> hist(x, main="Dichte von 1000 N(0,1) Zufallszahlen", freq=FALSE,
+ ylab="Dichte", xlim=c(-5, 5), ylim=c(0, 0.45))
> curve(dnorm, from=-5, to=5, add=TRUE, lty=2, lwd=3)
> legend(2, 0.4, legend=c("empirisch", "theoretisch"), lty=c(1,2))
> qqnorm(x) # Ein QQ-Plot
> qqline(x, lwd=2, col="red") # Eine rote Linie für die ideale
# Normalverteilung
# Beispiel für Regression
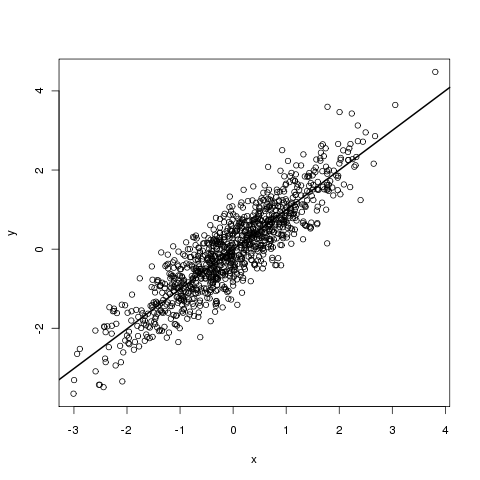
> y <- x + rnorm(x, sd=0.5) # Rauschen auf der y-Variablen
> plot(x,y)
> reg <- lm(y ~ x) # lineare Regression und speichern in reg
> summary(reg) # Zusammenfassung der Regression
> abline(reg, lwd=2) # einzeichnen der Regression
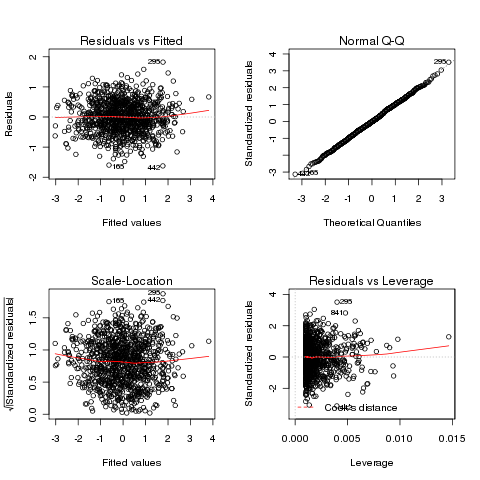
> par(mfrow=c(2,2)) # Grafik in 2x2 Subplots unterteilen
> plot(reg) # 4 verschiedene Plots zur Regression
Erklärung der Befehle
- set.seed() Für die Reproduzierbarkeit von Zufallszahlen lässt sich bei R mit diesem Befehl ein Startwert für den Pseudo-Zufallsgenerator setzen.
- rnorm() Erzeugt Normalverteilte Zufallszahlen.
- curve() Eine Funktion zum Zeichnen von Funktionen. Der optionale Parameter add=TRUE fügt die Kurve in die bestehende Grafik ein. lwd=3 Setzt die Linienbreite auf 3 lty=2 Setzt den Linientyp auf gestrichelt
- legend() Funktion vom Erzeugen von Legenden. Die Koordinaten der Position der Legende wird mit den ersten beiden Argumenten angegeben (x,y). Mit legend=c("1", "2") wird ein Vektor c() mit dem Text für die Legende übergeben. Mit lty=c(1,2) wird das Aussehen der Linien in der Legende bestimmt. Nutzen Sie auch die Hilfe ?legend
- qqnorm() Erzeugt einen \( QQ \)-Plot, wo die empirischen und die theoretischen Quartile einer Standardnormalverteilung \( N(0,1) \) gegeneinander aufgetragen werden. Wenn die Variable standardnormalverteilt ist, ergibt sich eine Linie, welche mit qqline() eingezeichnet wurde.
- lm() Erzeugt eine Regression, wobei das Modell der Regression als Formel angegeben werden muss. Dabei trennt die Tilde "~" die Zielvariable auf der linken Seite (abhängige Variable) vom Modell für die unabhängige Variable.
-
summary() Zusammenfassung der Regression:
Call: lm(formula = y ~ x) Residuals: Min 1Q Median 3Q Max -1.624218 -0.335981 -0.006877 0.377696 1.822160 Coefficients: Estimate Std. Error t value Pr(>|t|) (Intercept) -0.008093 0.016452 -0.492 0.623 x 1.003216 0.015904 63.078 <2e-16 *** --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 Residual standard error: 0.5202 on 998 degrees of freedom Multiple R-squared: 0.7995, Adjusted R-squared: 0.7993 F-statistic: 3979 on 1 and 998 DF, p-value: < 2.2e-16Es wird eine Zusammenfassung zur Verteilung der Residuen angegeben. Außerdem wird die Signifikanz der geschätzten Koeffizienten ausgegeben inklusive verschiedener Fehlerwerte für die Güte der Regression. - abline() Hierbei handelt es sich um eine Low-Level Funktion zum Zeichnen von "intelligenten" Linien. Da es sich nicht um eine eigene High-Level Funktion handelt, muss hier auch kein add=TRUE angegeben werden, da es sich hier nicht um eine eigenständige Grafik handelt.
- par(mfrow=c(2,2)) Bei dem Befehl par() handelt es sich um einen mächtigen Befehl, um alle Grafik-Parameter für Abstand, Ränder zu ändern. Mit mfrow=c(2,2) wird die Grafik in 2x2 Untergrafiken aufgeteilt. Siehe auch die Hilfe ?par für alle möglichen Einstellungsmöglichkeiten.
- plot(reg) Die Funktion plot() ist sehr vielseitig und besitzt verschiedene Möglichkeiten, verschiedenste Daten darzustellen. So ist es auch möglich, sich verschiedene Grafiken zu einer Regression ausgeben zu lassen.
Verteilungen
R besitzt als Programmiersprache für Statistik natürlich sämtliche Verteilungen als festen Bestandteil der Sprache. Dabei gibt es immer eine Funktion für die entsprechende Verteilung, welcher ein Buchstabe vorangestellt werden muss, je nachdem, was man benötigt:
- d (density) für den Wert der Dichtefunktion
- p (probability) für den Wert der entsprechenden Verteilungsfunktion
- r (random) Ziehen von Zufallszahlen
- q (quantiles) für die Berechnung von Quantilen
So ist die Funktion für die Normalverteilung _norm(), wobei _ durch den entsprechenden Buchstaben ersetzt werden muss. Zum Beispiel erzeugt rnorm(100, sd=0.5) 100 Zufallszahlen aus einer Normalverteilung mit Standardabweichung \( \sigma=0.5\)

Eine weitere nützliche Funktion zum Erzeugen von Stichproben ist sample() Die Funktion entspricht dabei dem Ziehen von Stichproben aus einer Urne. Das folgende Beispiel entspricht dem 200 maligen Würfeln mit einem fairen Würfel:
> wuerfel <- sample(c(1:6), 200, replace=TRUE, prob=rep(1/6, 6)) > wuerfel
Der allgemeine Aufruf ist dabei sample(x, size, replace=,prob=), wobei
- x ein Vektor mit den verschiedenen Elementen ist, aus denen gezogen werden soll. Im Beispiel mit dem Würfel sind das die Zahlen von 1 bis 6: c(1:6)
- size Gibt die Größe der zu ziehenden Stichprobe an. Im Beispiel wird 200 mal "gewürfelt"
- replace= gibt mit einem logischen Operator (TRUE/FALSE) an, ob es sich um Ziehen mit zurücklegen handelt
- prob= benötigt einen Vektor mit den entsprechenden Wahrscheinlichkeiten für die Elemente im Vektor x, aus denen gezogen wird. Im Beispiel mit dem Würfel wirf mit der Funktion rep(x,n) der Wert 1/6 6 Mal wiederholt, für jede der 6 Seiten des Würfels.