- Phasenbegriff: ist ein homogener Bereich
- thermodynamische Freiheitsgrade: Anzahl der Variablen, die ein System in Gleichgewicht vollständig beschreiben
Bsp. Wasser: \( FG=1-1+2=2 \) \( \rightarrow \) Es werden 2 Variablen benötigt, um Wasser thermodynamisch vollständig zu beschreiben: \( U=U(S,p) \) oder \( U=U(T,V)\)
Bsp. Wasser+Eis: \( FG=1-2+2=1 \) \( \rightarrow \) nur eine Variable ist frei wählbar. Im Experiment legt eine Druckänderung die Temperatur fest, bei der beide Phasen nebeneinander vorliegen können.
Anwendung: Phasenseparation an Membranen
- 1. Effekt: Wechselwirkungen ändern sich
- 2. Effekt: Entropieänderung -- Einschränkung der Bewegungsfreiräume reduziert die Entropie
Boltzmann Entropieformel
\[ S=k * \ln(W) \] ( \(k = \) Boltzmannkonstante) und ( \(W = \) Kombinationsmöglichkeiten) Ein Beispiel für die Anzahl der Kombinationmöglichkeiten \( (W)\): das Vermischen von 9Mol und 16Mol von 2 Flüssigkeiten: \( W=\frac{(9+16)!}{9!*16!} \) Ab einer ausreichend großen Molekühlanzahl kann die Stirling-Näherung verwendet werden:
Stirling-Näherung
\[ \Delta S \approx -k \left\lbrace N_A \ln(X_A)+N_B \ln(X_B) \right\rbrace \] ( \(N = \) Teilenanzahl: \( N=n*N \) und ( \(X = \) Molenbruch: \( X = \frac{n}{\sum n_i}\) ) Wenn man sich nicht für die Entropieänderung interessiert, dann kann die Lage des Gleichgewichts mittels \( \underline{\Delta G} \) charakterisiert werden.
Bestimmungsmethoden für \( \Delta G \)
Im biologischen Zusammenhang oder im Labor kann man \( \Delta T = 0 \) annehmen. Daraus ergibt sich eine alternative Gleichung: \[ \begin{eqnarray*} G &=& H - TS \\ dG &=& dH - d(TS) \rightarrow dG=dH -TdS -SdT \\ \mbox{es gilt \( \Delta T =0\)} \\ \mathbf{\Delta G} &\mathbf{=}& \mathbf{\Delta H - T\Delta S} \end{eqnarray*} \]
indirekte Messung von \( \Delta H \) und \( \Delta S\)
Vorteile: Anspruchslos: kein Labeln, kein Fixieren, automatisiert, viele Informationen, funktioniert immer bei ausreichend Probe
Nachteile: \( mg\)-Mengen erforderlich, sehr rauschanfällig da sehr sensibel, Messbereich für GG-Konstante auf \( 10^3<k<10^{12}
Isothermales-Titrations-Kalorimeter (ITC)
- Kalorimeter wird mittels Thermostat auf konstanter Temperatur gehalten.
- Ligand und Rezeptor werden zur Reaktion gebracht \( \rightarrow \) als Ergebnis steigt die Temperatur und man erhält \( \Delta H \) pro Tropfen Ligand
- danach sinkt die Temperatur wieder auf Thermostatwert
- der Anstieg der Temperatur pro Tropfen Ligand nimmt mit jedem Tropfen ab, bis alle Rezeptoren mit Liganden gebunden sind
- Ergebnisse: \( \Delta H\), \( \Delta S\); GG-Konstante; Anzahl der Bindungsstellen zwischen Ligand und Rezeptormolekühl
Differential-Scanning-Calorimetry
- für Phasenübegänge und Proteinfaltung
- Probe und Referenz werden kontinuierlich erwärmt \( \rightarrow \) Reaktionen in der Probe führen zu Abweichungen der Heizkurve mit der der Referenz
indirekte Messung der GG-Konstanten \( (K)\)
\[ K=\frac{[RC]}{[R][C]} = e^{\left(- \frac{\Delta G}{RT}\right)} \]
Gleichverteilungssatz
Wärme wird in Bewegung gespeichert
-
Gleichverteilungssatz jeder Freiheitsgrad\footnote{mechanische Freiheitsgrade = ''Bewegungsmöglichkeiten''} speichert die gleiche Wärmemenge \( \left(\frac{1}{2}kT\right)\)
Möglichkeiten: Rotation, Schwingung, Translationsrichtungen
Bei einer chemischen Reaktion \( A + B \rightarrow AB \) gehen Freiheitsgrade verloren und pro Freiheitsgrad wird \( \frac{1}{2}kT \) Energie frei. Zusätzlich zur Bindungsenergie kommt es noch zu Wärme- und Entropieänderungen.
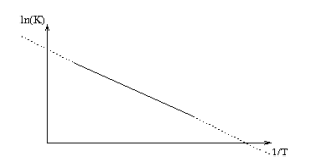
Van't-Hoff-Plot
Mit Hilfe des Van't-Hoff-Plots lässt sich nach einer Regression aus den gemessenen Werten für die Gleichgewichtskonstante (\(K\) ) und die Temperatur (\( T \) ) die gesuchten Größen \( \Delta G_0\), \( \Delta H_0 \) und \( \Delta S_0 \) berechnen. \[ \Delta G_0 = -RT \ln{K} = \Delta H_0 -T\Delta S_0 \] Die Gleichung wird nach dem Prinzip von Van't-Hoff linearisiert \[ \begin{eqnarray*} \ln{K} &=& - \frac{\Delta H_0}{R}*\frac{1}{T} + \frac{\Delta S_0}{R} \\ y &=& m x + n \\ y &=& \ln{K} \\ m &=& - \frac{\Delta H_0}{R} \\ x &=& \frac{1}{T} \\ n &=& \frac{\Delta S_0}{R} \end{eqnarray*} \]
Gleichgewichts-Dialyse:
- Reaktion durch eine semipermeable Membran: Rezeptor durch die Membran eingesperrt, der Ligand kann frei diffundieren.
- bekannt sind die Gesamtmenge an Rezeptor und Ligand \( [L_{ges}]\), \( [R_{ges}]\)
- gesucht: \( K=\frac{[RL]}{[L]*[R]}\)
- gemessen wird die \( [L] \) im GG
- lösen des Gleichungssystems: \[ \begin{eqnarray*} L_{ges}&=&RL+L\\ R_{ges}&=&RL+R \mbox{(Stoffmengen)} \end{eqnarray*} \]
- Vorteile: extrem einfach, preiswert
- Nachteile: hohe Konzentrationen erforderlich, einer der Bindungspartner darf nicht permeable sein, die Membran kann selbst Rezeptor oder Ligand unspezifisch binden
Flugzeit-Massenspektrometrie
- ein Ion wird im elektr. Feld zwischen 2 Elektroden beschleunigt
- Ion verlässt Elektrodenraum und fliegt weiter zum Detektor
- Messgröße: \( \Delta t \) für die Strecke bis zum Detektor
Energiezufuhr des Ions: \( E_z=q*U \) (\(q = \) Ladung des Ions) und (\(U = \) Beschleunigungsspannung) Kinetische Energie des Ions beim Austritt: \( E_k = \frac{m}{2} v^2\) \[ \begin{eqnarray*} v = \frac{d}{dt} \rightarrow &\Delta t =& \sqrt{\frac{1}{2U}}*d*\sqrt{\frac{m}{q}} \\ &=& \underbrace{\sqrt{\frac{1}{2Ue}}*d}_{Konstante} * \sqrt{\frac{m}{z}} \end{eqnarray*} \]
-
Massenspektrometrie von Proteinen:
- Proteine sind keine Ionen! \( \rightarrow \) fehlende Ladungen
- Lösung:Matrixmethode (MALDI)
- Einbetten in Matrixmaterial und anschließendes Verdampfen und Photooxidieren der Matrixmolekühle, bei der Oxidation entstehende Ladungen werden auf das Protein übertragen
- Elektrospray-Verfahren
- Proteinlösung wird durch geladene Kapillare gepresst, bei Verdampfen des Lösungsmittels gehen Ladungen auf Proteine über
- Vorteil: sehr genau, schnell, viele Anwendungen
- Nachteil: kaum für Membranproteine anwendbar, unphysiologische Einbettungsmedien
Fluoreszenz-Depolarisation
- polarisiertes Licht mit einer bestimmten Schwingungsebene \( \rightarrow \) Molekühl fluoresziert nur, wenn es sich zufällig in der richtigen Lage für Absorption befindet
- emittiert Fluoreszenzlicht in einer bestimmten Polarisationsebene
- kleiner fluoreszierender Ligand rotiert schneller als der größere Komplex
- Drehung führt zur Änderung der Polarisationsebene des Angestrahlten Lichts (Depolarisation) \( \rightarrow \) Unterscheidung der Molekülgröße möglich
- bestimmen der GG-Konstanten
- Vorteil: sensitiv, funktioniert auch in vivo, kleine Tropfmengen sind ausreichend
- Nachteil: Labeling notwendig
- Zeitskala: Absorption: \( 10^{-15}sek\); Fluoreszenz: \( 10^{-8}sek\); Molekülrotation: \( 10^{-10}-10^{-12}sek\)
Surface Plasmon-Resonanz
- Glasprisma wird an der Oberseite mit einem Goldfilm bedampft \( \rightarrow \) Anker für Proteine
- über Thio-Gruppen werden Rezeptorproteine verankert
- Laserstrahl wird an der Goldschicht reflektiert (Reflexion ist von der Schichtdicke abhängig)
- Ligand wird über die Rezeptoren eingeströmt und kann Bindung eingehen
- Lichtschwächung des Lasers steigt durch höheren Brechungsindex und stehende Wellen am Goldfilm
- Lichtschwächung ergibt Belegungsdichte und GG-Konzentrationen
- Vorteil: Goldspots können sehr klein sein \( \rightarrow \) abscannen von großen Arrays; hohe Auflösung kleinster Konzentrationen, keine Markierung der Bindungspartner nötig, Messung der Bindungskinetik möglich
- Nachteil: ein Bindungspartner muss chemisch am Goldfilm immobilisiert werden, unspezifische Bindungen auf der Goldschicht wirken störend
Absorptionsspektroskopie
- messen der Extinktion abhängig von der Wellenlänge \( \lambda \) \[ E(\lambda)=\log \left(\frac{I_0}{I}\right) \]
- Anwenden des Beer-Lamber-Gesetzes: \[ c = \frac{E(\lambda)}{\epsilon * d} \] mit dem Extinktonskoeffizienten ( \(\epsilon\) ) und der Küvettendicke ( \(d\) )
- zur Bestimmung der Konzentration von 2 Stoffen muss bei 2 Wellenlängen gemessen werden: \[ \begin{eqnarray*} E_{\lambda_1} &=& [A]*\underbrace{\epsilon_{A\lambda_1}d}_{\mbox{bekannt}} + [B]*\underbrace{\epsilon_{B\lambda_1}d}_{\mbox{bekannt}} \\ E_{\lambda_2} &=& [A]*\underbrace{\epsilon_{A\lambda_2}d}_{\mbox{bekannt}} + [B]*\underbrace{\epsilon_{B\lambda_2}d}_{\mbox{bekannt}} \end{eqnarray*} \]